联 系 人:吉恩特客服
手 机:136-0866-9917(微信同号)
地 址:河南省洛阳市高新区火炬创业园
Biotechnology Journal在线发表了华中农业大学张献龙教授领衔的棉花团队在植物学主流杂志题为“Whole genome sequencing reveals rare off-target mutations and considerable inherent genetic or/and somaclonal variations in CRISPR/Cas9-edited cotton plants” 的研究论文,该文利用高通量测序的方法,详细评估了CRISPR/Cas9 系统在棉花基因编辑过程中的脱靶效应。通过对野生型对照、阴性再生植株、Cas9编辑的阳性植株进行全基因测序,结果证明了CRISPR/Cas9编辑后代中存在数千个SNP/Indels 变异, 这些遗传变异来源于同一亲本的后代个体间的差异或者组织培养过程中的体细胞变异,其数量远远大于CRISPR脱靶位点的数量(4000多个预测位点中,只检测到4个脱靶位点),表明CRISPR/Cas9系统在多倍体植物基因组编辑具有高度特异性,脱靶率极低。
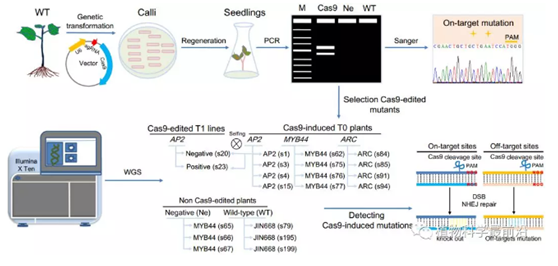
基因组编辑技术是当前生命科学研究的前沿领域,CRISPR/Cas9系统已用于治愈人类遗传病、实现细胞个性化治疗、开发新药、作物遗传改良等领域。CRISPR/Cas9技术依赖于Cas9核酸酶在gRNA指导下在PAM位点的上游3个碱基的位点切割目标DNA(On-target),但也会切割非靶标的位点(与sgRNA靶标位点序列相似,且具有PAM位点),这就造成所谓的Off-taget(脱靶),从而引起不可控的突变。因此,CRISPR技术存在的脱靶效应(Off-target effect)风险是影响CRISPR技术能否广泛应用的主要***因素,如何正确评估及降低脱靶效应是当前亟待解决的问题。目前对于脱靶检测方法主要有:1)依赖于核酸酶高灵敏度全基因方法;2)计算程序预测可能的脱靶位点,然后利用全基因组测序(WGS)检测脱靶位点是否发生突变。2017年,美国一个科研团队在Nature Methods发表的一遍关于小鼠基因编辑脱靶效应评估文章,他们推测CRISPR/Cas9可以引起数百个脱靶突变,该研究在学术界造成极大的轰动,也引起人们的广泛担忧。一家***的基因编辑的上市公司因为这篇论文的影响股票大跌12%。然而,很多科研工作者(也包括我们团队)在详细分析这个报道的相关数据后发现这项研究存在重大的缺陷:只有两只被CRISPR/Cas9编辑的小鼠用于实验,样本太少;而且这两只编辑的小鼠与野生型遗传背景不纯合,因此多少学者认为这篇文章中作者声称的所谓“脱靶”突变并不是由Cas9导致,而是样本本身存在的遗传差异造成 。2018年3月份该论文已经撤稿,来自基因编辑领域的多位***学者发表评论文章,指出这篇研究缺乏足够的对照、两只小鼠的遗传背景不同等诸多不足。关于CRISPR脱靶效应,在动物的基因编辑中有很已经有比较系统的报道,而关于植物脱靶效应在拟南芥、水稻等二倍体模式植物中有少量报道,几乎没有检测到任何非靶标突变。而这些研究都有以下不足:1)仅选择部分脱靶标位点进行深度测序,不是全基因组水平。2)全基因组测序对照太少,忽略了母体不同单株背景突变3)忽略了转基因过程中体细胞变异等。因此在植物中开展脱靶效应的深度评估,具有重要的理论和实践意义。
相比二倍体植物而言,棉花是异源四倍体作物,基因组大小在2.5Gb,包含两个亚基因组,靶位点编辑比较复杂,多拷贝数的同源基因或基因家族理论上可能会增加脱靶的可能性。本研究中利用CRISPR/Cas9系统对14个Cas9编辑植株(3个靶标基因的6个sgRNA位点),再加上3 株野生型 (WT)和3个再生群体中的阴性单株 (Ne,经历过组织培养过程,但不含有转基因成分) 作为对照进行35×测序深度进行全基因组测序。 研究结果表明,通过比较野生型和阴性对照,每个 Cas9 编辑的单株后代有 4188 ~ 6404 的 SNPs和 312 ~ 745 indels 变异,进一步对这些变异位点的侧翼序列分析,与靶位点没有同源性,绝大多数位点都缺少PAM位点。因此,我们认为这些变异位点来源于受体材料的不同后代(同一品种不同单株)之间的存在遗传差异以及棉花组织培养过程中的大量存在的体细胞无性系变异。 我们在4413个通过软件预测的潜在脱靶位点中仅检测到4个发生了真实的脱靶突变,并用Sanger测序证实。 同时,我们还通过对受体材料JIN668和TM-1参考基因组(用于设计sgRNA的基因组)遗传变异进行分析,检测到61个因为不同材料间的遗传变异可以产生新的脱靶位点,这些结果表明在设计sgRNA时要高度关注转基因母体基因组与参考基因组的遗传变异,利用计算程序减小脱靶风险性,同时我们建议在做基因编辑时候,***好能获得受体母本的基因组信息用于设计sgRNA,而不是用其它非受体材料的基因组信息。
这篇报道是2017年棉花团队建立***基因编辑体系的基础上(王鹏程、金双侠、张献龙等,High efficient multi-sites genome editing in allotetraploid cotton using CRISPR/Cas9 system, Plant Biotechnology Journal, 的后续系列报道, 这两个研究充分说明了该团队建立的基因编辑系统,具有极高的编辑效率(平均85%的编辑效率)和高度特异性,完全可以满足棉花功能基因组的研究需求。